[パッケージ紹介] csvファイルを超高速読込み vroomパッケージ
はじめに
超高速でreadr::read_csv()より速い
Rに限らずですが、データを扱う仕事をしていると巨大なデータを読み込むことがちょいくちょくあります。
私の経験上ファイルサイズが10MBを超えてくると、どのようにファイルを読み込むか、 どのように読み込んだファイルを処理するか、気を配らないととんでもない時間のロスにつながる恐れがあると思います。
今回紹介する{vroom}パッケージは、ファイルの読み込みに特化したパッケージです。 {vroom}パッケージは正直使ったことが無かったのですが、巨大なファイルの読み込みにかかる速度を既存の方法と比較してみたところ、vroom::vroom()を使った方が極めて高速であることが分かりました。
Snitch
最後にはベンチマーク結果も紹介します!
data.table::fread()とどっちが速いかは文字列の多さ次第
結論から言うと、文字列が多いほどvroomの方が早くなります。 逆に、数値データが多い場合にはdata.table::fread()の方が速いです。
これは公式のベンチマーク結果でも書かれており、 vroomが「文字列を毎回読み込んではメモリに格納しているわけではなく、インデックスだけを保存して後で参照している」 という仕様を持つことに由来します(ムズカシイ🤯)
私は今までほぼ使ってこなかったのですが、data.table::fread()も高速にデータを読み込むことのできる関数です。 今回は紹介しませんが、数値データが大量にあるファイルならdata.table::fread()を使う、というように使い分けるとなおよいでしょう。
比較的新しいパッケージである
最初に登場したのが2019年だそうです(リリース時のtidyverseブログより)。これだけ新しいパッケージがtidyverseの中枢に登場するのはかなり珍しいです。パッケージの開発もかなり盛んに行われているようですし、今後tidyverse界では存在感を放ちそうです。
基本的な使い方
読み込み
vroom::vroom()で読み込んだファイルはtibbleになって返ってきます。使い方は簡単です。
Hide
vroom::vroom("textfile.csv")このように、ファイルパスを指定するだけです。それぞれの列が文字列なのか数値なのか、 区切り文字がタブなのかカンマなのか、などは全て自動で判断してくれます(もちろん指定することも可能)。
書き込み
ファイル書き込みも出来ます。
Hide
mtcars %>%
vroom::vroom_write("mtcars.tsv")特に指定しない限りタブ区切りファイルとして出力されてしまいます。 csvにするには区切り文字(deliminator)を指定する必要があります。
mtcars %>%
vroom::vroom_write("mtcars.csv", delim = ",")csvにしたい時は一手間面倒ですが、公式のベンチマーク結果を見るとreadr::write_csv()よりも高速なようです。
少し応用的な使い方
web上のファイルも読み込める
vroomはウェブ上のcsv(カンマ区切り)やtsv(タブ区切り)ファイルを直接読み込むことができます。 厚生労働省が公開しているCOVID-19オープンデータ から、「新規陽性者数の推移(日別)」というファイルを読み込んでみます。
pacman::p_load(tidyverse)
vroom::vroom("https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv")Snitch
実はread.csv()関数も、readr::read_csv()関数も同じようにweb上のファイルを直接読み込むことができるんですけどね。
列を指定して読み込む
データフレームを扱う際には使用しない列が大元のデータには含まれていて、使う列だけを残したい、ということが非常によくあります。 readr::read_csv()などのreadrパッケージでファイルを読み込んだ際には、dplyr::select()関数を組み合わせて使うことで これが実現できます。
この方法は非常に有名で、一般的だとおもいます。私も数年来これで生きてきました。
Hide
read_csv("https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv") %>%
select(Date, ALL, Tokyo, Kanagawa)vroomでは、select()するステップをvroom関数内で指定することができます。
vroom::vroom("https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv",
col_select = c(Date, ALL, Tokyo, Kanagawa))また、列選択ではstarts_with, ends_withのメソッドが使えるようです。 まるでtidyselectの機能が適用されているかのようですが、どうもvroomで定義された関数で実装されていて挙動はtidyselectのそれとは異なるようです。
vroom::vroom("https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv",
col_select = starts_with("K")) この方法は一見するとメモリ管理上有利(全部のデータを保持すること無く、必要な列だけが読み込まれる)に思えますが、 実際にはvroom関数内で一度全データを読み込んでから列選択が行われているため、select()関数を使う場合とでコンピューターにかかる負担は同じです。
そのためこれは好みの問題で、美しい、書きやすい、覚えやすい方を選択すれば良いのかなと思います。
多数のファイルを結合して読み込む
次に紹介するのは、複数のファイルを一度に結合しつつ読み込む方法です。
これは以下のpurrr::map_dfr()に代わる方法として用意されているのだと思いますが、正直どっちを使うかは好みだと思います。
vroom以外の方法:purrr::map_dfr()を使う
まず、vroomを使わない方法です。
fs::dir_ls("test_data", glob = "*.csv") %>%
purrr::map_dfr(function(x) {
vroom::vroom(x) %>%
mutate(date = as.character(date))
})行っている操作を簡単に説明しておくと、fs::dir_ls()でtest_dataフォルダ以下にあるcsvファイルを全てリストアップしています。 今回は10個のテストデータを用意しています。fs::dir_ls()も今回知った関数でしたが、組込関数のlist.files()よりもかゆいところに手が届く感じでとても使いやすいと感じました!
fs::dir_ls("test_data", glob = "*.csv")
# test_data/2022-03-01.csv test_data/2022-03-02.csv test_data/2022-03-03.csv
# test_data/2022-03-04.csv test_data/2022-03-05.csv test_data/2022-03-06.csv
# test_data/2022-03-07.csv test_data/2022-03-08.csv test_data/2022-03-09.csv
# test_data/2022-03-10.csv これらを無名関数(vroom::vroom()で読み込み)に投げて、結果は全て結合された状態で返ってきます。結合された状態になるか否かはmap_dfr()を使うか、map()を使うかなどで変わってきます。
vroom::vroom()を使う方法
次に、vroomで一気に読み込むパターンです。初めて使ってみましたが、これはかなり簡単です!
先の例とは違って「vroomが面倒ごとを見えないところでうまく処理してくれている」ようなコードですので、 初心者の方は自分が何をやっているか分からなくなりやすい点に注意が必要です。
fs::dir_ls("test_data", glob = "*.csv") %>%
vroom::vroom()この方式で読み込む際に注意しなければならないのは、列の型が思ったとおりに読み込まれていなかった場合です。(日付型のつもりが文字列だった、数値のつもりが文字列だった等)
ダーティーな列名を修正する
Snitch
若干マニアックな内容なので、Rやtidyverseをある程度理解していないと混乱するかも。難しかったら読み飛ばしてOKです。
tidyverseではtidyselectというルールで列名を指定し、原則列名にはクオート("とか'のこと)を付けません。 select()関数で例を示すと、以下の通りです。
iris %>%
select(Sepal.Length, Sepal.Width)# 以下も同じ様に動作するが、本来の意図からは少々ずれている
# 昔は動作しなかった
iris %>%
select("Sepal.Length", "Sepal.Width")この場合に面倒なのが、列名が指定しにくいものになっている場合です。
参考例として以下のようなファイルを作ってみました。
Hide
vroom::vroom("dirty_data.tsv")
## A tibble: 3 × 4
# Participant ID Sample ID header 0_data
# <dbl> <chr> <chr> <chr>
#1 2 Taro 太 none
#2 5 Jiro 次 none
#3 6 Subro 三 none このデータはいくつかの罠が潜んでいます。
まず、最初の二つの列は空白が含まれるので、次のようにしなければ指定できません。
vroom::vroom("dirty_data.tsv") %>%
select(Participant ID,Sample ID)# または先の例
vroom::vroom("dirty_data.tsv") %>%
select("Participant ID","Sample ID")次のheaderという列は実は” header”という表記になっていましたが、読み込む段階で自動的に解消されています。
最後の”0_data”も数字から始まるため、同じような指定の仕方が必要です。
vroomだけではなくreadrの読み込み関数でも使える操作ですが、janitorパッケージと組み合わせると列名が良い感じに変更されます。
vroom::vroom("dirty_data.tsv",
.name_repair = janitor::make_clean_names)便利だけど覚えられるかはちょっと微妙…
速度計測
公式もベンチマークをしていますが、私の環境でもやってみました。 ベンチマーク方法はy__mattuさんのコードを参考にさせていただきました。
HMP2データ
Human Microbiome Project2 にて取得されたサンプルのメタデータを例にしてみます。
pacman::p_load(microbenchmark)
pacman::p_load(tidyverse)
hmp2_csv <- "/hdd50t/share/dl_data/IBDMDB/metadata/hmp2_metadata.csv"
compare_read <-
microbenchmark(
"read.csv()" = read.csv(hmp2_csv),
"readr::read_csv()" = read_csv(hmp2_csv),
"data.table::fread()" = data.table::fread(hmp2_csv),
"vroom::vroom()" = vroom::vroom(hmp2_csv)
)
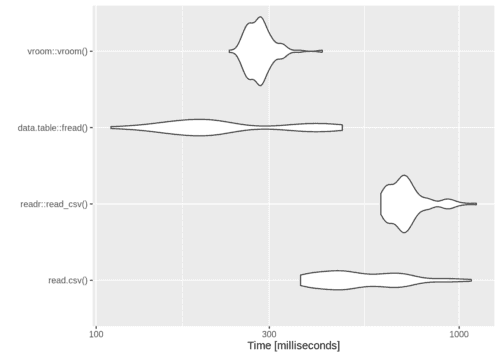
autoplot(compare_read)結果:vroomとdata.table::fread()が速い
結果としてはvroomがread_csv()よりも遙かに速い事が分かりました!私はread_csv()をメインで使っていたため、 これだけで業務改善です!
しかし、vroomとdata.table::fread()を比べると、若干fread()の方が速そうです。(計測値にバラツキがあるのはやや気になりますが・・・・
GTDBデータ
GeneTaxonomyDataBase(GTDB)のバクテリアメタデータでもベンチマークしてみます。
gtdb_tsv <- "/work2/oodake/GTDB/release_202/bac120_metadata_r202.tsv"
compare_read_2 <-
microbenchmark(
"read.csv()" = read.delim(gtdb_tsv, sep = "\t"),
"readr::read_csv()" = read_tsv(gtdb_tsv),
"data.table::fread()" = data.table::fread(gtdb_tsv),
"vroom::vroom()" = vroom::vroom(gtdb_tsv)
)
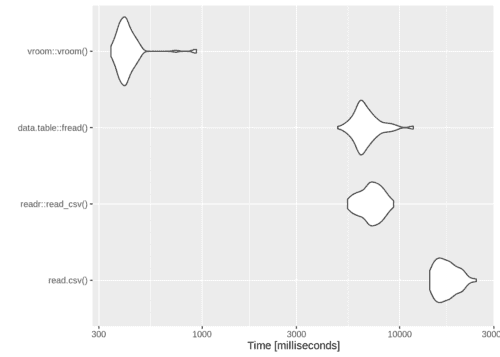
autoplot(compare_read_2)GTDBデータを使ったベンチマーク
結果:vroomの圧勝
このデータに関してはvroomが圧倒的に速かったようです。先ほどのデータの場合と見比べると全体的な傾向がやや異なっているので、データの性質に依る部分が大きそうです。
しかし組み込み関数のread.csv()よりも50倍近く速いなんて・・・すごすぎる
まとめ
今回はvroomパッケージを使ったファイルの読み込み方法をご紹介しました!まさかこんなに速いとは・・・最新のtidyverse系パッケージも、なんだかんだ技術の進歩が継続的にあることを実感させられました。
csvやtsvといった、一般的なデータ形式の読み込みでは既存の手法よりもかなり有利になりそうですので、私も今後多用していきたいと思います!
- vroom::vroom(“path/to/file.csv”)で高速読込ができる
- 比較的新しいパッケージで、tidyverseの一部である
- 複数ファイルの一括読み込み・結合は他の関数にないオリジナル機能
- readr::read_csv()と使用感はかなり似ている
- readr::read_csv()より高速
それではまた!👋